
2021 至今 深圳湾实验室 系统与物理生物学研究所 资深研究员、副所长
2013-2021 澳大利亚格里菲斯大学 终身正教授
2006-2013 美国印第安纳普渡大学 终身正教授
2004-2006 美国纽约州立大学布法罗分校 终身副教授
2000-2004 美国纽约州立大学布法罗分校 助理教授
1995-2000 美国哈佛大学 Martin Karplus组 博士后
1994-1995 美国北卡州立大学 Carol Hall组博士后
1994-1994 美国纽约州立大学石溪分校 George Stell组 博士后
1990-1993 美国加州应用物理化学实验室 科学家,实验室主任
1985-1990 纽约州立大学石溪分校 化学物理博士
1979-1984 中国科学技术大学 化学物理学士
周耀旗课题组的研究主要是围绕着RNA和蛋白质的序列、结构及功能之间关系的基础研究以及生物高分子的设计、递送、及药物开发的应用研究。课题组研究的特色是干湿结合(结构生物信息计算、AI人工智能深度学习与现代高通量、自动定向进化生物技术相结合)来实现对序列、结构及功能之间关系的深刻理解,从而达到生物高分子在多方面应用的目的,其中包括为精准医疗服务的针对性药物设计、递送和个性化生物标志物的检测。目前的具体项目包括、但不局限于:
· 后AlphaFold时代的蛋白质结构和功能预测
· 蛋白质设计和自动定向进化
· RNA结构预测,RNA语言模型
· RNA靶向递送
首创利用浅层与深度学习技术实现连续主链二面角预测,使端到端蛋白质结构预测成为可能。这一进展为诺奖工作AlphaFold 2 初步攻克这个分子生物领域长达60年的重大难题奠定了直接基础(图一)。
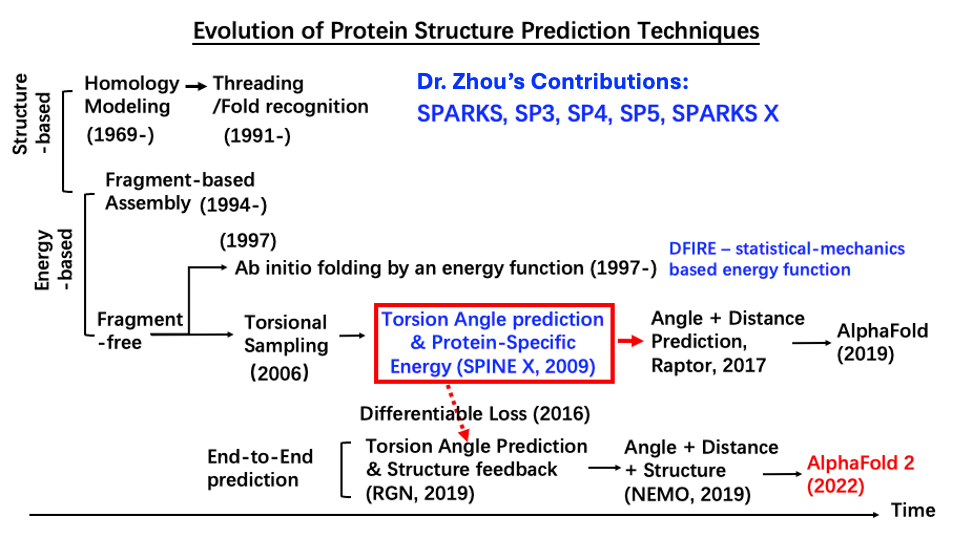
图一
率先开发人工智能驱动的蛋白质序列设计方法SPIN/SPIN2,实现30–34%的序列恢复率(图二)。该成果被国际权威期刊Current Opinion in Chemical Biology(Ovchinnikov与Huang,2021)评价为机器学习应用于蛋白质设计的起点,标志着该领域研究范式从传统基于能量的方法向人工智能驱动的根本性转变。如今这一范式已主导蛋白质设计领域,推动治疗性蛋白与工业酶设计的革命性进展。
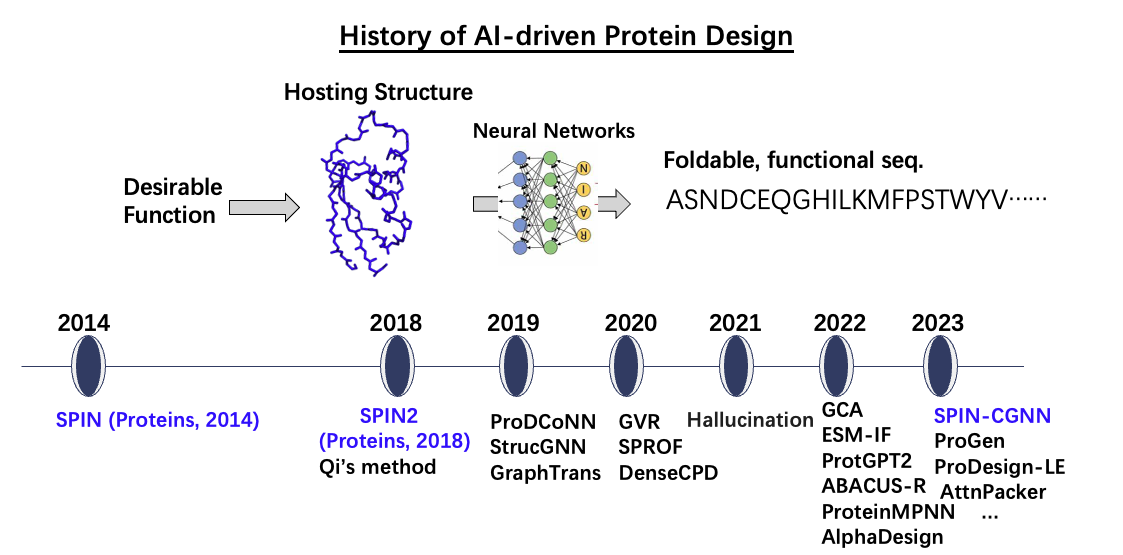
图二
开发了一种新型统计势能RNA-BRiQ,实现了原子级RNA结构优化。该技术赋能AIChemy-RNA2斩获第十五届结构预测关键评估(CASP15,2022年)RNA结构预测冠军。
· 全球前2%顶尖科学家终身科学影响力排行榜和年度科学影响力排行榜,中国高被引学者生物学榜
· 20,000+ 引用,H因子79(谷歌学者)
· 国际预测比赛第一名:CASP6基于模板蛋白质结构(2004), CAGI细胞扩增(2013), CAID蛋白质内在无序结构(2019), CASP15 RNA结构(2022)
· 畅销书《出发:不断走出舒适区的科研生活之旅》作者
· 砺博生物科学创始人,靶向RNA小分子药物平台
· 国际杂志《核酸研究》中国区唯一编委
1. Y. Zhou, T. Litfin, and J. Zhan, 3=1+2: How the Divide Conquered de Novo Protein Structure Prediction and What’s Next? National Science Review, 10:nwad259 (2023).
2. P. Xiong, R. Wu, J. Zhan, and Y. Zhou, “Pairing a high-resolution statistical potential with a nucleobase-centric sampling algorithm for improving RNA model refinement.”, Nature Communications, 12,2777 (2021).
3. J. Singh, J. Hanson, K. Paliwal, and Y. Zhou, RNA secondary structure prediction using an ensemble of two-dimensional deep neural networks and transfer learning, Nature Communications 10, 5407 (2019).
4. Z. Zhang, P. Xiong, T. Zhang, J. Wang, J. Zhan, and Y. Zhou, Accurate inference of the full base-pairing structure of RNA by deep mutational scanning and covariation-induced deviation of activity, Nucleic Acids Research, 48:1451-1465 (2020).
5. J. Zhan, H. Jia, E. A. Semchenko, Y. Bian, A. M. Zhou, Z. Li, Y. Yang, J. Wang, S. Sarkar, M. Totsika, H. Blanchard, F. E.-C. Jen, Q. Ye, T. Haselhorst, M. P. Jennings, K. L. Seib, and Y. Zhou, Self-derived structure-disrupting peptides targeting methionine aminopeptidase in pathogenic bacteria; a new strategy to generate antimicrobial peptides, FASEB J. , 33: 2095–2104 (2019).
6. Z. Li, Y. Yang, J. Zhan, L. Dai and Y. Zhou, Energy Functions in De Novo Protein Design: Current Challenges and Future Prospects, Ann. Rev. Biophysics 42, 315-335 (2013).