糖在生命活动中扮演着能量供给、细胞信号传导、免疫识别等至关重要的角色,其多维功能依赖于其独特的立体结构。然而,与核酸和蛋白质相比,糖的结构与功能研究相对滞后。这主要是由于单糖分子类型多样,具有多个手性中心,形成聚糖时糖苷键连接方式复杂多变,且结构高度柔性,使得其高分辨率三维结构的解析异常困难,严重限制了我们对其结构与功能的深入理解。
近年来,颜宁团队提出了 “CryoSeek(酷寻)”研究策略,在没有先验知识的情况下,将冷冻电镜作为面向未知生物大分子的发现工具。因此,CryoSeek策略或代表一种“前向结构生物学”研究,即以结构为先导的生物学发现范式。在前期研究中,颜宁团队结合冷冻电镜技术、AI辅助的自动建模以及生物信息学分析,报道了来自清华大学荷塘环境样品的一种纤维蛋白结构TLP-1,并对其来源和潜在功能进行了推测。随后的研究进一步鉴定出一种新型糖纤维结构TLP-4,其核心是由四肽重复序列组成的线性多肽,外围被厚厚的糖链包裹。四肽重复片段包含一个保守的3,4-二羟脯氨酸 (3,4-dihydroxyproline, DiHyp) ,其3-OH与4-OH均高度O-糖基化。与DiHyp相邻的位置,还存在一个保守的O-糖基化修饰的丝氨酸或苏氨酸。这些研究成果,不仅揭示了聚糖在生物大分子结构组装过程中的重要作用,也为天然糖质的发现与结构解析提供了新思路 [1,2,3]。
然而,在利用冷冻电镜解析结构时,会面临一个根本性挑战:冷冻电镜成像丢失了结构的“绝对手性”信息,这导致最终重构得到的三维密度图存在“镜像颠倒”的两种可能。对于蛋白质,科学家可利用其α-螺旋均为右旋这一先验知识来校正。但糖质纤维主要由糖构成,不含α-螺旋,且自然界同时存在D型和L型糖,因此即使达到原子分辨率,也无法直接判断绝对手性。这一不确定性如同失去了“地图的南北方向”,使得构建正确的原子模型寸步难行。
此前,解决该问题的经典方法是“倾转对”成像技术,但它需要预先知道样品的参考结构,并对同一区域进行两次成像。对于从自然水体等环境中直接获取的、可能包含上百种不同糖质纤维结构的高异质性样品而言,传统方法几乎无法应用。
北京时间2025年10月1日,颜宁团队、胡名旭团队及其合作者在预印本网站 BioRxiv 发布两项最新研究成果,论文标题分别为“基于CryoSeek 策略鉴定具有多样组成与结构组装的糖纤维”(CryoSeek identification of glycofibrils with diverse compositions and structural assemblies)和“冷冻电子显微学中自然来源糖质纤维的绝对手性确定”(Absolute hand determination of glycofibrils from natural sources in cryo-EM)。


延续“荷糖月色”研究计划,团队在后续的数据处理中解析了另外五种全新的糖纤维结构,揭示了糖纤维结构在天然环境中的多样性以及糖质在结构组装过程中的关键作用。与此同时,为解决糖纤维绝对手性判定这一难题,研究团队合作开发了名为Ahaha的新方法,该方法仅需在单角度倾斜的情况下进行常规的冷冻电镜成像,即可高效、准确地判定天然来源糖纤维的绝对手性,目前,该方法已开通在线服务(图1)。
图1. Ahaha的在线服务页面(https://cryoseek.org/ahaha)
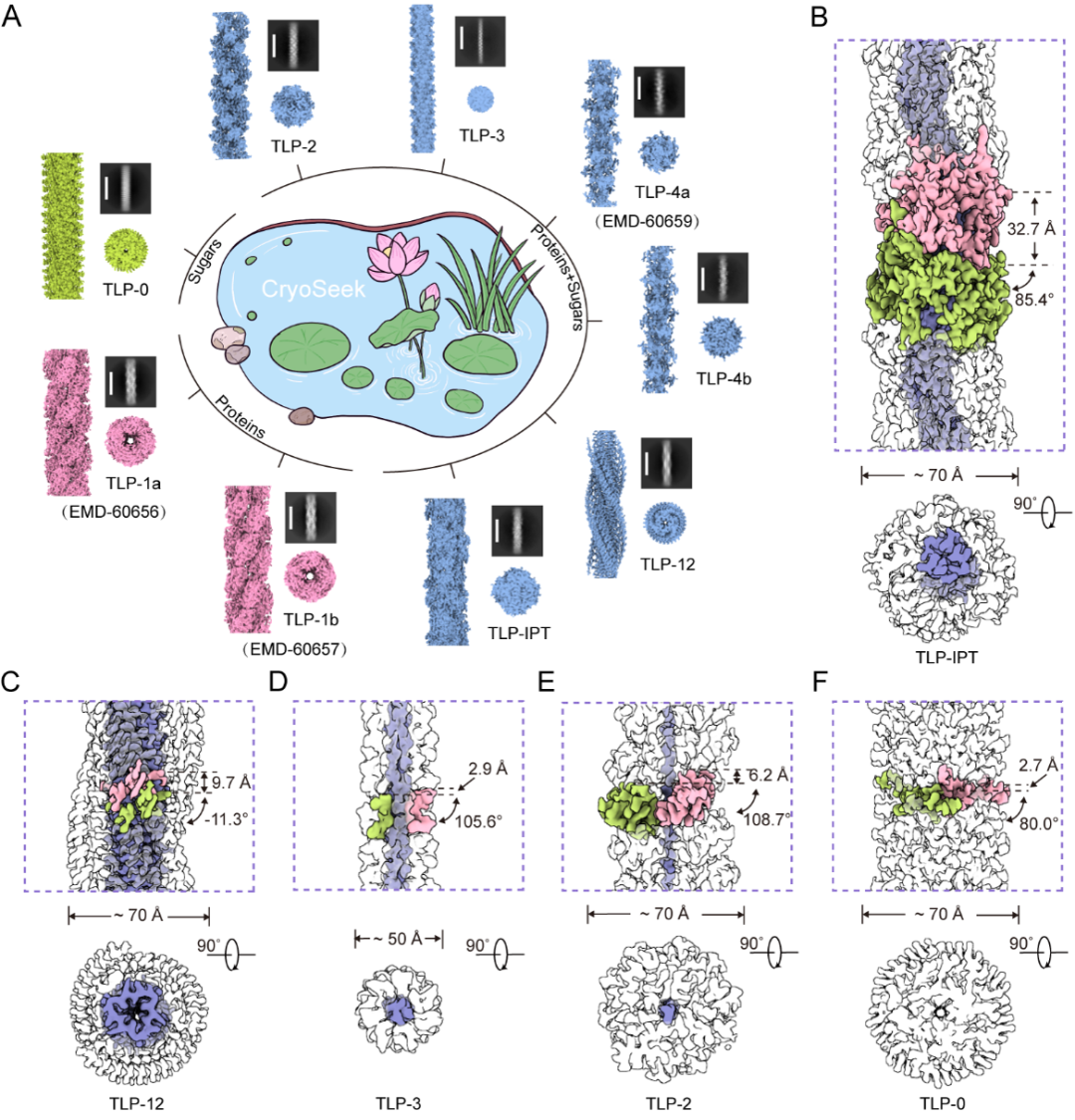
图2. 利用CryoSeek研究策略,在清华荷塘中鉴定出多种纤维结构。
图2A. 目前,在清华荷塘水样中共解析出9种高分辨率纤维结构,依据其蛋白组成可分为三种类型,分别为蛋白纤维(TLP-1a/b)、以蛋白为核心的糖纤维(TLP-2/3/4a/4b/12/IPT)和全部由糖组成的纤维(TLP-0);
图 2B-F. 最新报道的5种糖纤维结构的组成以及其螺旋参数。
新解析的五种糖纤维分别命名为TLP-IPT、TLP-12、TLP-3、TLP-2和TLP-0,其中名称前缀的TLP为清华大学荷塘的缩写(Tsinghua Lotus Pond),后缀则反映了各糖纤维中蛋白核心的特征。TLP-IPT是由连续IPT(Ig‑like/plexins/transcription factors)结构域构成可识别的蛋白核心,每个IPT结构域外围被13条糖链环绕;TLP-12是由三股高度重复的十二肽共同编织成三股平行β‑折叠带,外围覆盖两列螺旋糖脊;TLP-3和TLP-2的核心都是线性多肽,其中TLP-3由三股三肽重复序列互绞形成蛋白细芯,而TLP-2则是一条二肽重复序列构成,它们外圈都被厚厚的一层糖链包裹;TLP-0则完全由糖质组成,“0”表示其不含有蛋白组分。上述研究不仅揭示了天然环境中糖纤维的结构以及组成的多样性,也表明以CryoSeek策略的框架下,结合多学科研究手段,有望为糖质测序以及天然糖质三维结构的高通量、体系化研究提供新路径。
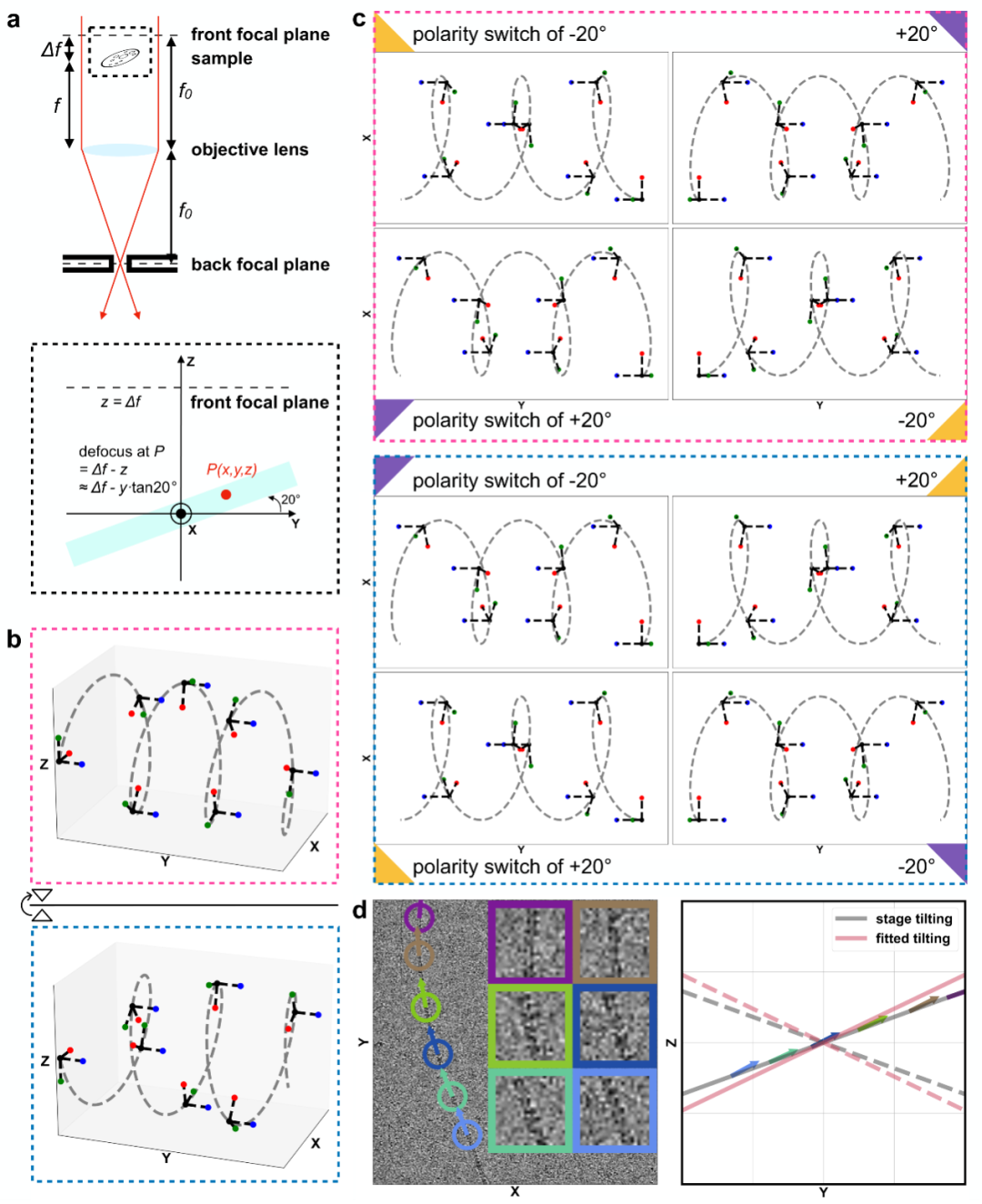
图3. Ahaha决定手性的原理示意图
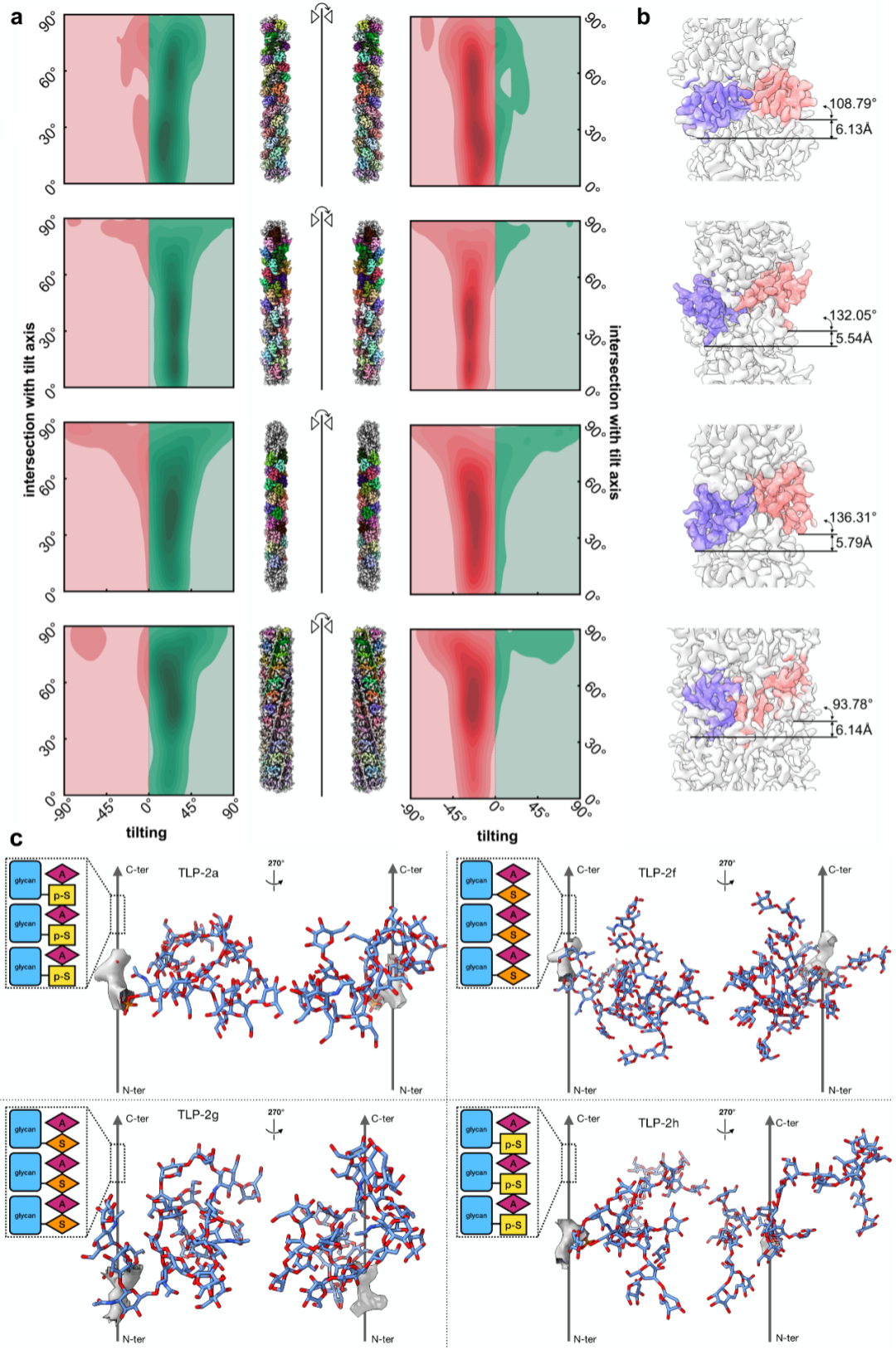
图4. 使用Ahaha决定四种自然来源糖质纤维的手性并搭建其原子模型。
图4a. Ahaha分析了四种糖质纤维的颗粒倾斜角的热力图绿色表示颗粒倾斜角与给定样品倾斜角一致,从而表示其三维结构手性正确,红色则表示相反从而手性错误。作为对照,使用了相反的螺旋转角(twist)参数与手性相反的初始模型进行螺旋精修时,Ahaha能够如预期地给出手性错误的结果。
图4b. 糖质纤维相邻两个螺旋单元,及螺旋对称参数。
图4c. 糖质纤维的原子模型。
深圳医学科学院、深圳湾实验室颜宁教授,清华大学生命学院助理研究员李张强, 和王彤彤博士为第一篇预印本论文的共同通讯作者。清华大学生命学院的李张强助理研究员、王彤彤博士、孙熠彤是该论文的第一作者。清华大学生命学院的徐魁博士、黄文泽博士、张强锋副教授、闫创业副教授,以及深圳医科院的胡名旭特聘研究员为该论文做出了重要贡献。深圳医学科学院特聘研究员胡名旭,清华大学生命学院副教授王佳伟,深圳医学科学院、深圳湾实验室颜宁教授为第二篇预印本论文的共同通讯作者。深圳医学科学院张起博士是该文的第一作者。覃兰菊、王彤彤博士、李张强助理研究员、张毅琳、陈晟博士为研究工作做出了重要贡献。研究受到深圳医学科学院、国家自然科学基金、北京生物结构前沿研究中心资助。
参考文献:
[1] Wang, T., Li, Z., Xu, K., Huang, W., Huang, G., Zhang, Q. C., & Yan, N. (2024). CryoSeek: A strategy for bioentity discovery using cryoelectron microscopy. Proceedings of the National Academy of Sciences, 121(42), e2417046121.
[2] Wang, T., Huang, W., Xu, K., Sun, Y., Zhang, Q.C., Yan, C., Li, Z., & Yan, N. (2025). CryoSeek II: Cryo-EM analysis of glycofibrils from freshwater reveals well-structured glycans coating linear tetrapeptide repeats, Proceedings of the National Academy of Sciences, 122(1), e2423943122.
[3] Wang, T., Sun, Y., Li, Z., & Yan, N. (2024). The 8-nm spaghetti: well-structured glycans coating linear tetrapeptide repeats discovered from freshwater with CryoSeek. bioRxiv, 2024-12.