在传统的结构生物学研究范式中,研究者通常以感兴趣的,并且已知编码基因的生物大分子为研究对象,通过解析其三维结构,试图从分子层面揭示其功能与作用机制。然而,自然界中仍潜藏着数量庞大的未知生物大分子,它们可能并非由基因直接编码,或尚未被测序。颜宁团队另辟蹊径,尝试以冷冻电镜技术作为一种发现的工具,并提出CryoSeek研究策略,将冷冻电镜与人工智能建模、生物信息学分析等多学科技术相结合,使研究者能够在没有任何先验知识的情况下,从自然水体、环境样品乃至临床样本中直接发现并解析未知生物大分子的高分辨率结构,并以结构为基础进一步探索并阐明其功能。
在前期研究中,颜宁团队逐步建立并完善了CryoSeek这一结构生物学新范式。颜宁团队率先报道了来自清华大学荷塘环境样品的一类蛋白纤维结构TLP-1a和TLP-1b,并对其来源和潜在功能进行了推测。在此基础上,团队进一步鉴定出一种新型糖纤维结构TLP-4,其核心是由四肽重复序列组成的线性多肽,外围被厚厚的糖链包裹。并且后续的数据处理中又解析了另外五种全新的糖纤维结构,揭示了天然环境中糖化纤维的多样性,以及糖链在结构组装过程中的关键作用。同时,为解决糖纤维绝对手性判定的技术难题,颜宁团队与胡名旭团队合作开发了名为Ahaha的新方法。这些研究成果,不仅揭示了聚糖在生物大分子结构组装过程中的重要作用,也为天然复杂聚糖的发现与结构解析提供了新思路 [1,2,3,4]。
尽管CryoSeek研究策略具有显著的创新性,但其整体通量仍面临瓶颈,这主要体现在两个环节:图像处理和糖质建模。首先,自然样品中通常包含数十至上百种不同类型的纤维结构,其中许多成分的丰度极低。传统的冷冻电镜图像处理方法难以有效应对这种高度异质性,导致数据分析效率远低于数据采集速度。其次,即使获得了高分辨率的密度图,其精确模型的构建仍存在挑战。不同于可依赖预测算法或自动化搭建的蛋白质模型,糖残基的结构建模主要依赖人工的经验性识别。这一高度依赖人工的过程极大地限制了从密度图中解析糖结构的效率,因而构成了制约高通量糖结构生物学发展的第二个关键瓶颈。
北京时间2025年11月21日,颜宁团队、胡名旭团队及其合作者们在预印本网站浪淘沙公布最新成果,论文标题为“High-throughput cryo-EM characterization and automated model building of glycofibrils via CryoSeek”(通过CryoSeek实现糖质纤维的高通量冷冻电镜表征与自动化模型构建)。

图1:浪淘沙预印本
研究团队开发了两个新型方法以解决上述两个关键瓶颈:首先,研究团队引入了递归二分聚类(recursive bisection clustering,RBC)策略(图2),实现了对高度异质性的冷冻电镜数据的高效、并行处理,使图像分析通量得以匹配数据采集速度;其次,研究团队提出了人工智能驱动的EModelG 自动化建模框架(图3),能够精准识别糖残基位点并自动组装糖链,将糖质结构的建模从繁重的人工判别中解放出来。基于这两种方法,研究团队成功构建了一套高通量糖质纤维结构解析流程。利用该流程,研究团队系统性地分析了来自多个自然水样的样本,并解析了众多糖质纤维结构。为了整合与共享这些结构资源,研究团队初步搭建了CryoSeek数据库,为未来糖结构生物学的研究提供了宝贵的数据基础与方法支撑。
为解决CryoSeek策略中因样本高度异质性和目标低丰度导致的数据处理瓶颈,研究团队开发了递归二分聚类(RBC)这一高效图像处理方法。RBC采用自上而下的分裂式聚类策略:算法从螺旋识别得到的全体颗粒数据出发,通过逐层递归地二维分类将母节点颗粒不断一分为二,最终构建出一棵二叉树。这一设计使其具备两大关键优势:首先,随着递归深度增加,可并行处理的计算任务呈指数增长,极大提升了计算效率;其次,12层深度的RBC理论上可实现低至约0.24‰丰度目标的检测灵敏度。实际应用中,研究团队通过对桂林溶洞水样中1220万颗粒进行RBC处理,仅需1–2天即成功分离出18种丰度在0.14‰–4.9‰之间的纤维结构,充分证明了该方法在处理大规模、高异质冷冻电镜数据时的高效性与灵敏性。
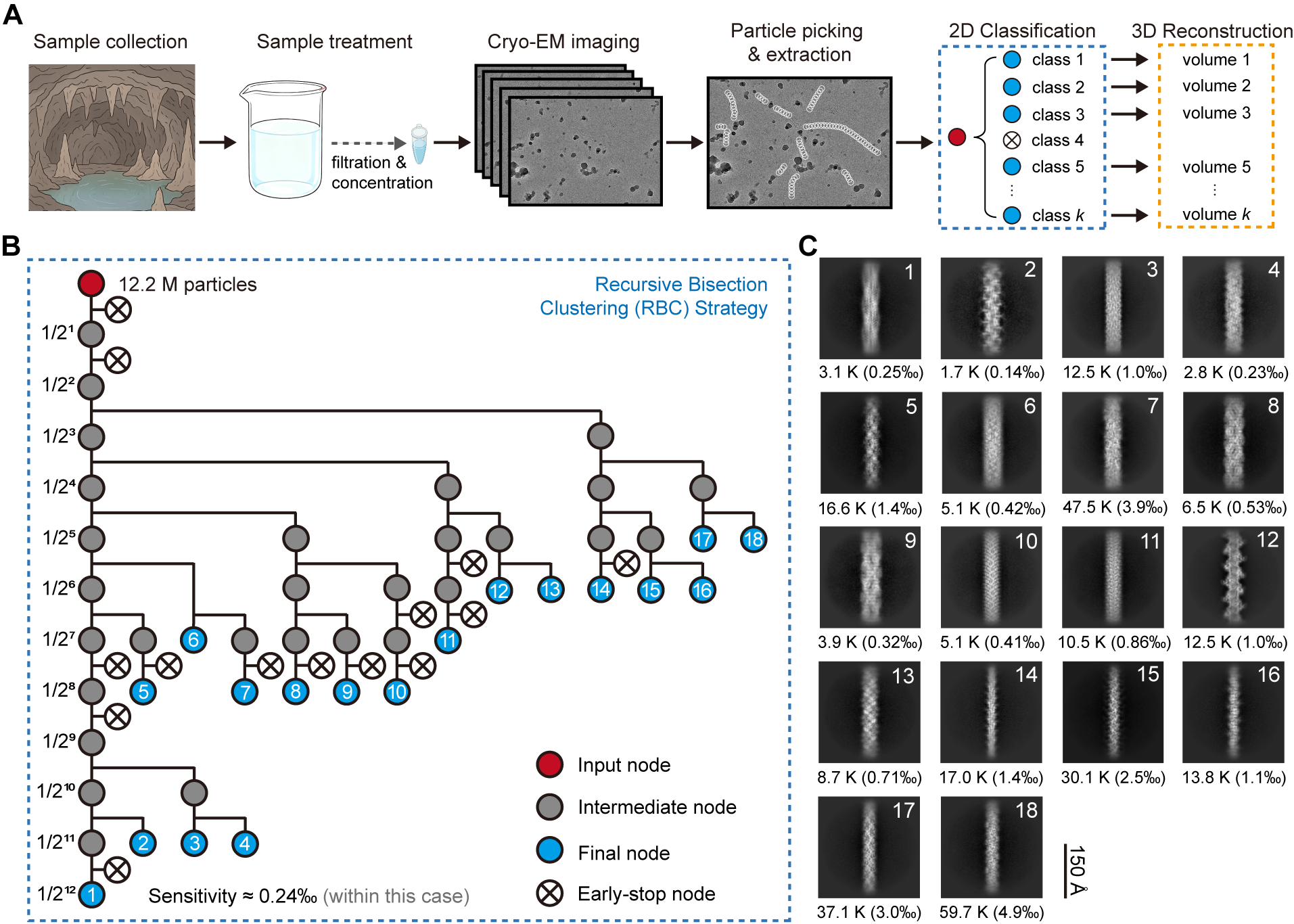
图2:利用递归二分聚类策略
从1220万颗粒中高效提取18种纤维结构
研究团队进一步开发了EModelG——首个可直接基于冷冻电镜密度图实现糖蛋白与糖链结构全自动建模的人工智能框架。该框架首先通过神经网络对密度图进行像素级解析,区分蛋白质与糖区域;随后自动构建蛋白质模型并提供糖基化位点作为锚点;在此基础上,EModelG通过SE(3)旋转采样单糖模板、梯度优化的密度拟合及糖苷键自动组装,实现从蛋白质表面延伸糖链的全程自动化建模。以糖蛋白GF-1L2R5(2.53 Å)为例,EModelG准确重建了完整糖链分支,其自动搭建的单糖环与实验密度高度吻合,且与人工模型高度一致。为CryoSeek流程中糖质结构的高通量、标准化模型搭建奠定了关键技术基础。
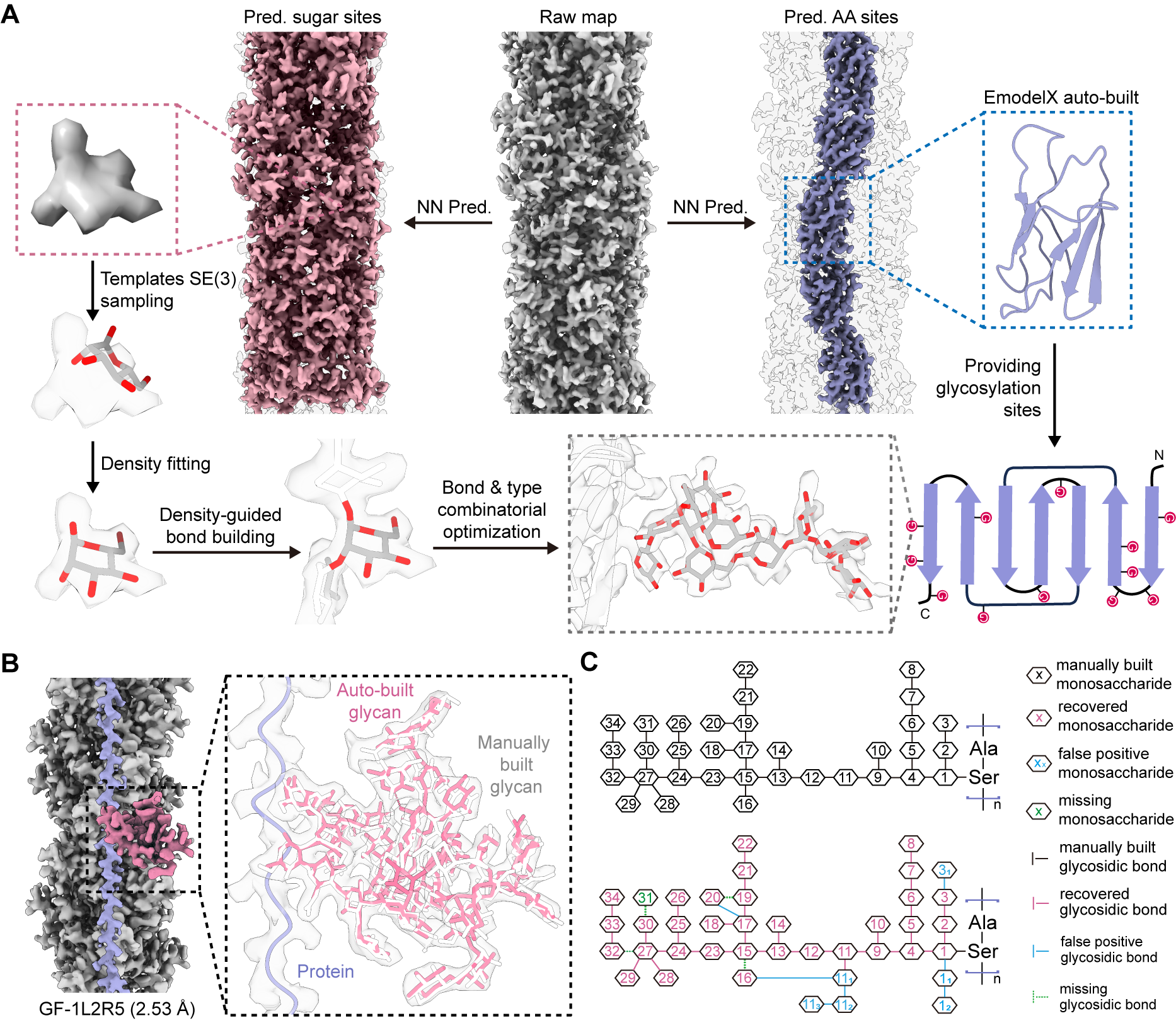
图3:自动化糖质模型搭建的人工智能框架EModelG
通过应用RBC策略和EModelG,研究团队成功将CryoSeek的数据处理能力提升至与数据采集通量相匹配的水平。从13个不同来源的水样中,共解析出126个三维螺旋结构,并从中归纳出50类具有代表性的纤维结构。为系统描述这些结构,研究团队建立了以分子组成和结构特征为基础的命名体系:根据组成分为蛋白纤维(PF,protein fibril)与糖质纤维(GF,glycofibril);进而依据蛋白结构域、线性肽重复单元或完全无蛋白(NP,no protein)等特征进行后缀细分。这套命名方案既满足了对新结构的包容性,也实现了对已有结构的系统化归类,为后续大规模糖质纤维结构研究建立了标准化基础。

图4: 50个具有代表性的三维螺旋结构
为了整合与共享这些结构资源,研究团队还搭建了CryoSeek数据库。CryoSeek数据库是一个集成了糖质纤维结构、电镜密度图及原始显微图像数据,并保留其间关联性的综合资源平台。该数据库不仅支持常规的数据检索与上传功能,还提供了一系列特色工具与服务:包括面向糖质纤维的绝对手性判定工具Ahaha、用于全球科研合作的社区交流平台,以及面向教学与培训的专项资源库,从而为糖结构生物学领域提供数据共享、方法支持与科研协作的一体化基础设施。
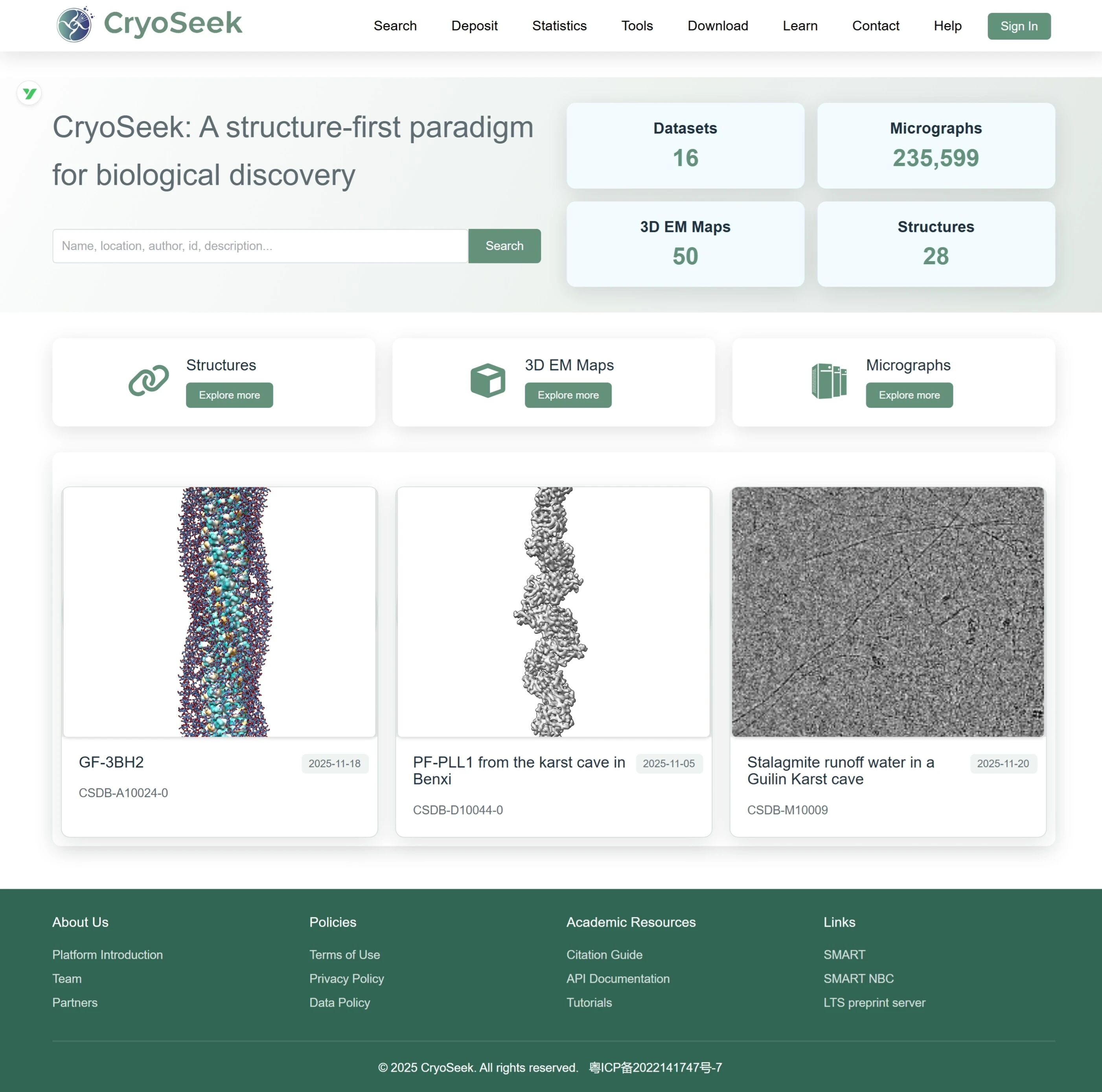
图5:CryoSeek数据库 (https://cryoseek.org.cn)
深圳医学科学院创始院长、深圳湾实验室主任颜宁、深圳医学科学院特聘研究员胡名旭、清华大学生命科学学院李张强博士为本文的共同通讯作者。深圳医学科学院特聘研究员胡名旭、清华大学生命科学学院陈晟博士、王彤彤博士、深圳医学科学院覃兰菊、张起博士为本文的共同第一作者。深圳医学科学院张毅琳、戈其珺为研究工作做出重要贡献。研究受到深圳医学科学院、国家自然科学基金、北京生物结构前沿研究中心资助。
参考文献:
[1] Wang, T., Li, Z., Xu, K., Huang, W., Huang, G., Zhang, Q. C., & Yan, N. (2024). CryoSeek: A strategy for bioentity discovery using cryoelectron microscopy. Proceedings of the National Academy of Sciences, 121(42), e2417046121.
[2] Wang, T., Huang, W., Xu, K., Sun, Y., Zhang, Q.C., Yan, C., Li, Z., & Yan, N. (2025). CryoSeek II: Cryo-EM analysis of glycofibrils from freshwater reveals well-structured glycans coating linear tetrapeptide repeats, Proceedings of the National Academy of Sciences, 122(1), e2423943122.
[3] Li, Z., Wang, T., Sun, Y., Xu, K., Huang, W., Zhang, Q.C., Yan, C., & Yan, N. (2025). CryoSeek identification of glycofibrils with diverse compositions and structural assemblies, bioRxiv, 2025.09.30.679562.
[4] Zhang, Q., Qin, L., Wang, T., Li, Z., Zhang, Y., Chen, S., Yan, N., Wang, J., & Hu, M. (2025). Absolute hand determination of glycofibrils from natural sources in cryo-EM, bioRxiv, 2025.09.30.679555.